文章信息
文章题目:Functional RNA splitting drove the evolutionary emergence of type V CRISPR-Cas systems from transposons
期刊:Cell
发表时间:2025 年 9 月 29 日
主要内容:中国科学院遗传与发育生物学研究所高彩霞研究团队,联合清华大学生命科学学院刘俊杰副教授、中国科学院动物研究所张勇研究员首次发现并定义了连接转座子与 CRISPR 之间长期缺失的关键进化中间体,命名为 TranC (Transposon-CRISPR intermediate),弥合了 CRISPR 进化历程中的缺口。研究揭示,驱动 TnpB 转座酶向 Cas12 系统演化的核心机制源于引导 RNA 的“功能性分裂”,而非蛋白质结构的根本性改变。这一发现不仅破解了 Cas12 起源的分子机制之谜,也首次以实验证据阐明了 RNA 层面的创新如何驱动复杂分子机器的进化进程。
原文链接:
https://doi.org/10.1016/j.cell.2025.09.004
使用TransGen产品:
2×TransStart® FastPfu PCR SuperMix (AS221)
TransDetect® PCR Mycoplasma Detection Kit (FM311)

研究背景
CRISPR-Cas 系统是原核生物的获得性免疫系统,Cas9 与 Cas12 效应蛋白已被开发为主流基因组编辑工具。研究表明,Cas12 由 IS200/605/607 转座子编码的 TnpB 多次独立演化而来,但转座子如何跃迁至免疫系统的分子机制仍缺关键证据。
文章概述
研究通过序列、结构、催化基序三重筛选,从原核基因组与宏基因组中捕获 146 个 TnpB 近邻蛋白,系统发育与 AlphaFold 预测锁定 6 个过渡家族,命名 TranC。它们与不同 TnpB 分支成姊妹群,代表 IS605/IS607 转座子多次独立孕育 Cas12 的演化路径。功能实验显示,TranC 同时保留祖先 reRNA 单导模式并启用 CRISPR 双 RNA(tracrRNA+crRNA)切割,人类细胞中 LaTranC 可兼用两种向导完成基因组编辑。冷冻电镜揭示,TranC 蛋白三维结构几乎与 TnpB 重叠,差异仅在 RNA:单一 reRNA 分裂为独立 tracrRNA 与 crRNA 模块,形成典型 CRISPR 双导构架;该“RNA 功能分裂”在其余支系亦被共变异预测普遍验证。进一步人工拆分 ISDra2 TnpB 的 reRNA 为嵌合双 RNA 后,原系统即获得阵列识别与切割能力,完成从转座子到免疫系统的角色转变。研究据此阐明,RNA 模块化创新而非蛋白重构,是驱动 CRISPR-Cas 多次独立起源的核心分子引擎,为定向设计微型精准核酸酶提供新思路。
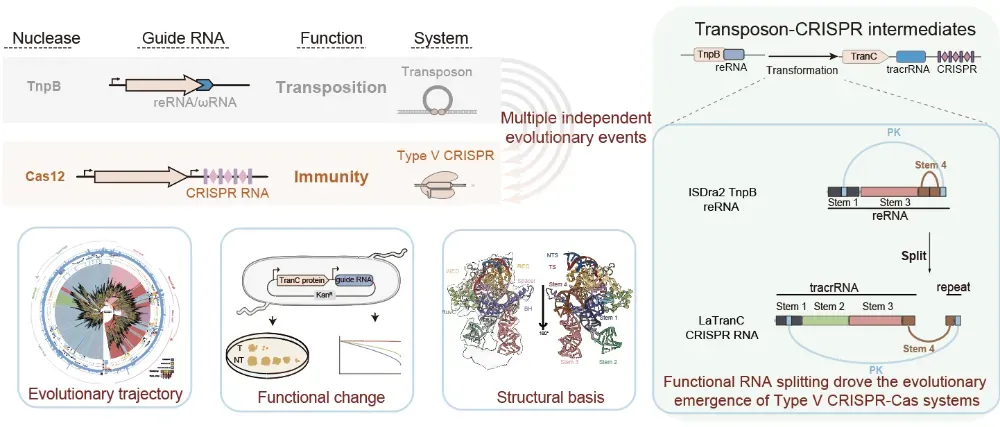
TranC系统的发现揭示了CRISPR起源的关键分子机制
全式金生物产品支撑
优质的试剂是科学研究的利器。全式金生物的核酸提取试剂(ER101)、PCR 试剂(AS221)、支原体检测试剂(FM311)助力本研究。产品自上市以来,凭借优异的性能,深受客户青睐,多次荣登知名期刊,助力科学研究。
EasyPure® RNA Kit (ER101)
本试剂盒适用于从培养的动物细胞、组织和大肠杆菌中快速提取总 RNA。样品被异硫氰酸胍裂解,DNA 被 DNase I 消化,RNA 与硅胶膜离心柱特异地结合。提取的总 RNA 纯度高,没有 DNA 和蛋白质污染,可用于 RT-PCR、qRT-PCR、芯片分析、Northern blot 等实验。
产品特点
• 操作简单时间短:30 分钟即可完成 RNA 的提取。
• 适用范围广:动物细胞、组织和大肠杆菌均可快速提取总 RNA。
• 纯度高:独特的设计可高效去除样本中的杂质。
• DNA 污染:有 DNase I消化步骤,去除 DNA 更彻底。
2×TransStart® FastPfu PCR SuperMix (AS221)
本产品含 TransStart® FastPfu DNA Polymerase、dNTPs 和优化的反应缓冲液,浓度为 2×,扩增效率强,扩增速度快,具有高保真性、高特异性。DNA 扩增时,只需加入模板、引物和水,使 SuperMix 溶液的浓度为 1× 即可进行反应。扩增产物为平端,可直接克隆于 pEASY®-Blunt 系列载体中。含 dye 版本扩增产物可直接点样电泳。
产品特点
• 快速:4 kb/min 快速扩增。
• 简单:2× 的 PCR 扩增 Mix 形式,加入引物、模板和水即可扩增;扩增完成后可直接电泳检测,无需额外加入 Loading Buffer。
• 热启动、特异性强。
• 扩增效率高:复杂模板、富含 GC/AT 的模板以及长片段也可以有效扩增。基因组 DNA 片段的扩增(≤15 kb),Plasmid DNA 片段扩增(≤20 kb)。
• 高保真:保真性是 EasyTaq® DNA Polymerase 的 54 倍。
TransDetect® PCR Mycoplasma Detection Kit (FM311)
本产品通过 PCR 检测的方法针对支原体 16S rRNA 保守序列设计特异引物,以细胞培养液为模板直接扩增支原体 DNA。可检出 54 种支原体,包括口腔支原体、肺炎支原体、猪鼻支原体、精氨酸支原体、发酵支原体、唾液支原体、梨支原体、人型支原体、莱氏无胆甾原体等。
产品特点
• 特异性强:仅扩增支原体 DNA,不扩增真核细胞及细菌 DNA。
• 操作简便:无需提取基因组 DNA,适合大批量样品。
• 快速灵敏:PCR 法检测,速度快、灵敏度高。
• 质控完善:含阳性对照与阴性对照,结果可靠。
使用 EasyPure® RNA Kit (ER101)产品发表的部分文章:
• Jin S, Zhu Z, Li Y, et al. Functional RNA splitting drove the evolutionary emergence of type V CRISPR-Cas systems from transposons[J]. Cell, 2025.(IF 42.50)
• Zhou Y, Zhu S, Cai C, et al. High-throughput screening of a CRISPR/Cas9 library for functional genomics in human cells[J]. Nature, 2014.(IF 41.46)
• Han X, Wang R, Zhou Y, et al. Mapping the mouse cell atlas by microwell-seq[J]. Cell, 2018.(IF 31.40)
• Liu M, Wang D, Qi C, et al. Brain ischemia causes systemic Notch1 activity in endothelial cells to drive atherosclerosis[J]. Immunity, 2024.(IF 25.50)
• Lv Z, Chen L, Chen P, et al. Clearance of β-amyloid and synapses by the optogenetic depolarization of microglia is complement selective[J]. Neuron, 2024.(IF 16.20)
• Chen W, Ma J, Wu Z, et al. Cas12n nucleases, early evolutionary intermediates of type V CRISPR, comprise a distinct family of miniature genome editors[J]. Molecular Cell, 2023.(IF 16.00)
• Wu Z, Zhang Y, Yu H, et al. Programmed genome editing by a miniature CRISPR-Cas12f nuclease[J]. Nature chemical biology, 2021.(IF 15.04)
• Fu J, Wu S, Bao N, et al. A Universal Strategy of Anti-Tumor mRNA Vaccine by Harnessing“Off-the-Shelf”Immunity[J]. Advanced Science,2025.(IF 14.10)
• Wang Y, Luo W, Huang L, et al. A novel lncRNA linc-AhRA negatively regulates innate antiviral response in murine microglia upon neurotropic herpesvirus infection[J]. Theranostics, 2021.(IF 11.55)
• Zhao K, Wang L, Qiu D, et al. PSW1, an LRR receptor kinase, regulates pod size in peanut[J]. Plant Biotechnology Journal, 2023.(IF 10.10)
使用2×TransStart® FastPfu PCR SuperMix (AS221)产品发表的部分文章:
• Jin S, Zhu Z, Li Y, et al. Functional RNA splitting drove the evolutionary emergence of type V CRISPR-Cas systems from transposons[J]. Cell, 2025.(IF 42.50)
• Song R, Guo P, Ren X, et al. A novel polypeptide CAPG-171aa encoded by circCAPG plays a critical role in triple-negative breast cancer[J]. Molecular Cancer, 2023.(IF 37.30)
• Jin S, Lin Q, Gao Q, et al. Optimized prime editing in monocot plants using PlantPegDesigner and engineered plant prime editors (ePPEs)[J]. Nature Protocols, 2023.(IF 17.02)
• Li X, Zhang S, Wang C, et al. Efficient in situ epitope tagging of rice genes by nuclease-mediated prime editing[J]. The Plant Cell, 2025. (IF 11.60)
• Li X, Zhang S, Wang C, et al. Efficient in situ epitope tagging of rice genes by nuclease-mediated prime editing[J]. The Plant Cell, 2025.(IF 10.00)
• Wang Y, Wang Z, Chen Y, et al. A highly efficient CRISPR-Cas9-based genome engineering platform in Acinetobacter baumannii to understand the H2O2-sensing mechanism of OxyR[J]. Cell Chemical Biology, 2019.(IF 6.70)
使用 TransDetect® PCR Mycoplasma Detection Kit (FM311)产品发表的部分文章:
• Lei Z, Meng H, Liu L, et al. Mitochondrial base editor induces substantial nuclear off-target mutations[J]. Nature, 2022.(IF 69.50)
• Huang J, Lin Q, Fei H, et al. Discovery of deaminase functions by structure-based protein clustering[J]. Cell, 2023.(IF 65.00)
• Liu J L, Yan X Q, Wu H, et al. RNA codon expansion via programmable pseudouridine editing and decoding[J]. Nature, 2025.(IF 50.50)
• Xu D, Zhao H, Jin M, et al. Modulating TRADD to restore cellular homeostasis and inhibit apoptosis[J]. Nature, 2020.(IF 42.78)
• Jin S, Zhu Z, Li Y, et al. Functional RNA splitting drove the evolutionary emergence of type V CRISPR-Cas systems from transposons[J]. Cell, 2025.(IF 42.50)
• Fei H Y, LI Y J, Liu Y J, et al. Advancing protein evolution with inverse folding models integrating structural and evolutionary constraints[J]. Cell, 2025.(IF 42.50)
• Sun C, Li H C, Liu Y J, et al. Iterative recombinase technologies for efficient and precise genome engineering across kilobase to megabase scales[J]. Cell, 2025.(IF 42.50)
• Bai X, Sun P, Wang X, et al. Structure and dynamics of the EGFR/HER2 heterodimer[J]. Cell Discovery, 2023.(IF 38.07)
• Wang D, Li X, Jiao D, et al. LCN2 secreted by tissue-infiltrating neutrophils induces the ferroptosis and wasting of adipose and muscle tissues in lung cancer cachexia[J]. Journal of Hematology & Oncology, 2023.(IF 28.50)
• Nian Z, Dou Y, Shen Y, et al. Interleukin-34-orchestrated tumor-associated macrophage reprogramming is required for tumor immune escape driven by p53 inactivation[J]. Immunity.2024.(IF 25.50)
• Li Z, Wang Z, Zhong C, et al. P53 upregulation by USP7-engaging molecular glues[J]. Science Bulletin, 2024.(IF 18.80)
• Xu J, Liang Y, Li N, et al. Clathrin-associated carriers enable recycling through a kiss-and-run mechanism[J]. Nature Cell Biology, 2024. (IF 17.30)
• Fan S, Qi M, Qi Q, et al. Targeting FAPα-positive lymph node metastatic tumor cells suppresses colorectal cancer metastasis[J]. Acta Pharmaceutica Sinica B, 2024.(IF 14.80)
• Wang J, An Z, Wu Z, et al. Spatial organization of PI3K-PI (3, 4, 5) P3-AKT signaling by focal adhesions[J]. Molecular Cell, 2024.(IF 14.50)
• Zhang W, Yin C, Qi L, et al. RFWD3 reprograms nucleotide metabolism through PHGDH to induce chemoresistance in osteosarcoma[J]. Advanced Science, 2025.(IF 14.10)
• Nian Z, Zheng X, Dou Y, et al. Rapamycin pretreatment rescues the bone marrow AML cell elimination capacity of CAR-T cells[J]. Clinical Cancer Research, 2021.(IF 12.50)
• Wang L, Wang Y, He X, et al. CD70-targeted iPSC-derived CAR-NK cells display potent function against tumors and alloreactive T cells[J]. Cell Reports Medicine, 2025.(IF 10.60)







