文章信息
文章题目:Iterative recombinase technologies for efficient and precise genome engineering across kilobase to megabase scales
期刊:Cell
发表时间:2025 年 8 月 4 日
主要内容:中国科学院遗传与发育生物学研究所高彩霞团队在 Cell 杂志上在线发表了题为“Iterative recombinase technologies for efficient and precise genome engineering across kilobase to megabase scales”的研究论文,系统报道了一种新型可编程的染色体水平大片段 DNA 精准操纵技术 PCE (Programmable Chromosome Engineering)。该技术在动植物中实现了从千碱基到兆碱基级别 DNA 的多种类型且精准无痕的编辑,显著提升了真核生物基因组的操纵尺度和能力。
原文链接:
https://doi.org/10.1016/j.cell.2025.07.011
使用TransGen产品:
TransDetect® PCR Mycoplasma Detection Kit (FM311)

背景介绍
基因组编辑技术作为生命科学领域的一项革命性突破,为基础研究和应用开发提供了强大的技术支撑。以 CRISPR 及其衍生技术为代表的基因编辑平台,通过向导 RNA 精确引导核酸酶靶向基因组特定位点,已广泛应用于特定碱基和短片段 DNA 的精准编辑。然而,大片段 DNA 编辑仍存在显著技术瓶颈,尤其是数千至数百万碱基规模的精准操纵仍是领域内亟待解决的核心难题。攻克这一技术壁垒将为基因组大尺度操作提供关键支持——包括基因关键区域的替换与修复、功能基因序列的完整插入、基因簇的删减或重定位,以及人工构建基因组结构变异等。这一突破不仅能推动遗传操作技术的革新,还将为疾病治疗和作物改良领域带来重大进展。理想的大片段 DNA 编辑工具应能实现染色体水平的多类型编辑,涵盖精准插入、替换、删除、倒位和易位等操作。但现有工具在编辑效率、操作尺度、精准度及功能多样性等方面仍存在明显不足。
文章概述
Cre-Lox 重组系统虽然具备染色体水平 DNA 编辑能力,但在实际应用中受到三个关键问题的制约:首先,Lox 位点的对称结构导致重组反应可逆,显著降低了目标编辑效率;其次,Cre 酶需要形成四聚体才能发挥作用,提升其活性的工程改造难度高;最后,重组后特异性位点残留,影响编辑产物的精确性。
研究团队针对现有技术瓶颈制定了分步突破策略。首先,开发了高通量重组位点快速改造平台,并提出不对称 Lox 位点设计原则,成功创制新型 Lox 变体,其在保持高效正向重组能力的同时,将可逆重组活性显著降低至接近阴性对照水平。其次,进一步利用自主研发的融合蛋白通用逆折叠模型、结构与进化约束信息的蛋白定向进化平台 AiCE,对 Cre 蛋白的多聚化界面进行精确改造,成功获得了重组活性提高 3.5 倍的工程化 Cre 蛋白变体。最后,创建并优化了重组酶的无痕编辑策略 Re-pegRNA,利用引导编辑器的高效编辑特性,通过设计特异性 pegRNA 对重组后残留的 Lox 位点进行“重引导编辑”(re-prime editing),将其精准替换为原有基因组序列。基于这些技术突破,研究团队成功开发了 PCE 和 RePCE 两大可编程染色体编辑系统,可对不同 Lox 位点的插入位置和方向进行灵活编程,实现从 kb 到 Mb 尺度的大片段 DNA 精准无痕操纵。实验验证表明,该系统在动植物细胞中可高效完成多种基因组操作,包括 18.8 kb 大片段 DNA 的定点整合、5 kb 序列的定向替换、12 Mb 的染色体倒位、4 Mb 的染色体删除及整条染色体的易位,并利用该技术创制了含 315 kb 精准倒位的抗除草剂水稻种质。
综上,该研究开发了新型染色体编辑系统 PCE,在动植物中实现了跨越 kb 到 Mb 级别的多类型染色体精准操纵。该工具不仅能实现多基因叠加,还可通过操控基因组结构变异,为作物性状改良和遗传疾病治疗开辟新路径。此外,该技术有望推动新型育种策略的发展,例如通过操纵遗传连锁、调控重组频率实现育性控制,以及消除连锁累赘,充分释放野生种质资源中优异等位基因的育种潜力。精准染色体编辑技术的突破将加速人工染色体构建,在合成生物学等新兴领域也有重要的应用前景。
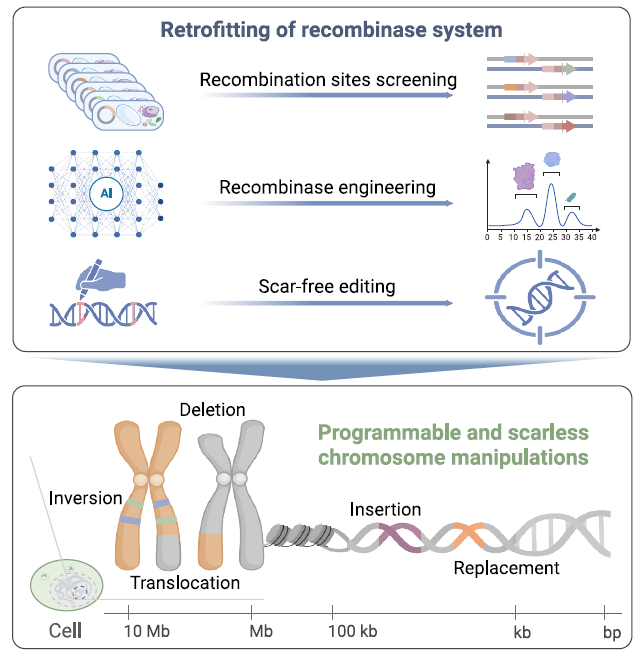
PCE 系统的开发和精准染色体编辑概述
全式金生物产品支撑
优质的试剂是科学研究的利器。全式金生物的支原体检测试剂盒(PCR法)(FM311)、氨苄青霉素(GG101)、卡那霉素(GG201)助力本研究。产品自上市以来,深受客户青睐,多次荣登知名期刊,助力科学研究。
TransDetect® PCR Mycoplasma Detection Kit (FM311)
一款通过 PCR 方法检测培养细胞等生物材料中的支原体,针对支原体 16S rRNA 序列保守区域设计特异引物,直接使用细胞培养液作为模板,特异性扩增支原体 DNA。
产品特点
• 检测灵敏度高、准确性好。
• 特异性强,只扩增支原体 DNA。
• 操作简便,无需提取基因组 DNA。
Ampicillin (GG101)
Ampicillin 为青霉素的衍生物,可通过干扰细菌的细胞壁合成来杀死生长细胞。其抗性机制为抗性基因 (Bla) 特异性表达外周质酶—β-内酰胺酶,该酶可切割抗生素的 β-内酰胺环。
Kanamycin (GG201)
卡那霉素 (kanamycin) 为一种杀菌剂,通过结合于 70S 核糖体,引起 mRNA 的错读。其抗性机制为抗性基因 (Kan) 特异性表达氨基糖苷磷酸转移酶,可修饰抗生素,抑制其与核糖体结合。
全式金生物的产品再度亮相 Cell 期刊,不仅是对全式金生物产品卓越品质与雄厚实力的有力见证,更是生动展现了全式金生物长期秉持的“品质高于一切,精品服务客户”核心理念。一直以来,全式金生物凭借对品质的执着追求和对创新的不懈探索,其产品已成为众多科研工作者信赖的得力助手。展望未来,我们将持续推出更多优质产品,期望携手更多科研领域的杰出人才,共同攀登科学高峰,书写科研创新的辉煌篇章。
使用 TransDetect® PCR Mycoplasma Detection Kit (FM311) 产品发表的部分文章:
• Sun C, Li H C, Liu Y J, et al. Iterative recombinase technologies for efficient and precise genome engineering across kilobase to megabase scales[J]. Cell, 2025.(IF 45.6)
• Liu J L, Yan X Q, Wu H, et al. RNA codon expansion via programmable pseudo uridine editing and decoding[J]. Nature, 2025.(IF 50.5)
• Fei H Y, LI Y J,Liu Y J, et al. Advancing protein evolution with inverse folding models integrating structural and evolutionary constraints[J]. Cell, 2025.(IF 45.6)
• Nian Z, Dou Y, Shen Y, et al. Interleukin-34-orchestrated tumor-associated macrophage reprogramming is required for tumor immune escape driven by p53 inactivation[J]. Immunity, 2024.(IF 25.5)
• Xu J, Liang Y, Li N, et al. Clathrin-associated carriers enable recycling through a kiss-and-run mechanism[J]. Nature Cell Biology, 2024.(IF 17.3)
• Wang J, An Z, Wu Z, et al. Spatial organization of PI3K-PI (3, 4, 5) P3-AKT signaling by focal adhesions[J]. Molecular Cell, 2024.(IF 14.5)
• Huang J, Lin Q, Fei H, et al. Discovery of deaminase functions by structure-based protein clustering[J]. Cell, 2023.(IF 65.00)
• Lei Z, Meng H, Liu L, et al. Mitochondrial base editor induces substantial nuclear off-target mutations[J]. Nature, 2022.(IF 69.50)
• Nian Z, Zheng X, Dou Y, et al. Rapamycin pretreatment rescues the bone marrow AML cell elimination capacity of CAR-T cells[J]. Clinical Cancer Research, 2021.(IF 11.4)
• Xu D, Zhao H, Jin M, et al. Modulating TRADD to restore cellular homeostasis and inhibit apoptosis[J]. Nature, 2020.(IF 50.5)
使用 Ampicillin (GG101) 产品发表的部分文章:
• Sun C, Li H C, Liu Y J, et al. Iterative recombinase technologies for efficient and precise genome engineering across kilobase to megabase scales[J]. Cell, 2025.(IF 45.6)
• Liu L, Zou L, Li K, et al. Template-independent genome editing in the Pcdh15av− 3j mouse, a model of human DFNB23 nonsyndromic deafness[J]. Cell Reports, 2022. (IF 9.995)
• Jin S, Fei H, Zhu Z, et al. Rationally designed APOBEC3B cytosine base editors with improved specificity[J]. Molecular cell, 2020.(IF 15.58)
使用 Kanamycin (GG201) 产品发表的部分文章:
• Sun C, Li H C, Liu Y J, et al. Iterative recombinase technologies for efficient and precise genome engineering across kilobase to megabase scales[J]. Cell, 2025.(IF 45.6)
• Gong T, Hu C Y, Zhang C Q, et al. Catalytic Mechanism of Gut Benzyl Ether Reductase for Efficient Bioconversion of Furofuran Lignans into Enterolignan Precursors[J]. Journal of Agricultural and Food Chemistry, 2025. (IF 6.2)
• Jin S, Fei H, Zhu Z, et al. Rationally designed APOBEC3B cytosine base editors with improved specificity[J]. Molecular cell, 2020.(IF 15.58)







