文章信息
文章题目:A cGAS-mediated mechanism in naked mole-rats potentiates DNA repair and delays aging
期刊:Science
发表时间:2025 年 10 月 9 日
主要内容:同济大学生命科学与技术学院/附属妇产科医院毛志勇教授团队在 Science 期刊发表题为“A cGAS-mediated mechanism in naked mole-rats potentiates DNA repair and delays aging”的研究论文,首次揭示裸鼹鼠通过 cGAS 蛋白的适应性演化,将人类细胞中的 DNA 修复抑制因子转化为修复增强因子,为抗衰老干预提供新靶点。
原文链接:
https://doi.org/10.1126/science.adp5056
使用TransGen产品:
Fast Mutagenesis System (FM111)

研究背景
DNA 修复能力是决定物种寿命的关键因素。裸鼹鼠(Heterocephalus glaber,Naked mole-rat)作为最长寿的啮齿类动物,其寿命可达约 40 年,表现出对多种疾病的显著抵抗能力。因此,揭示其健康维持的内在机制,已成为衰老研究领域的重要方向。已有研究指出,裸鼹鼠具有强大的基因组稳定性维持能力,然而其中所涉及的特异性分子调控机制仍有待阐明。深入解析裸鼹鼠独特的 DNA 修复机制,不仅能进一步揭示基因组稳定性与长寿之间的内在联系,也有望为开发延缓衰老、促进健康长寿的新干预策略提供关键理论依据。
文章概述
本研究发现,裸鼹鼠 cGAS 能够显著增强同源重组修复(HR)修复能力,且该功能独立于经典的 DNA 感应通路,这与该团队在 2018 年发现的人类 cGAS 在 DNA 损伤后入核抑制 HR 进而导致基因组不稳定的现象相反。为了理解这一现象背后的分子机制,研究团队利用 DNA 修复报告系统锁定了裸鼹鼠 cGAS 的 C 端结构域中 4 个进化特异的氨基酸(S463, E511, Y527, T530),发现这些位点的突变会导致修复功能丧失,而将其引入人类 cGAS 则可逆转其对 HR 的抑制。进一步研究发现,裸鼹鼠 cGAS 可在 DNA 损伤后比人类同源蛋白更持久地停留在染色质,而这是由于 4 个氨基酸变异降低了 cGAS 的 K48 泛素化修饰,削弱其与解离酶 P97 的互作,从而阻止其从损伤位点提前解离。并通过促进 FANCI 与 RAD50 的相互作用,加速 RAD51 的招募以增强 HR 修复。功能研究表明,表达裸鼹鼠 cGAS 可减缓辐射诱导的细胞衰老,而将裸鼹鼠 4 个位点引入人类 cGAS,可抵消其对细胞衰老的促进作用。在果蝇中表达裸鼹鼠 cGAS 可改善肠道功能、运动能力并延长寿命,而在小鼠中也有助于抵抗多器官衰老并延长寿命。这些结果共同揭示,裸鼹鼠 cGAS 有助于个体的健康增龄及长寿。
本研究首次揭示长寿物种通过 DNA 修复抑制因子 cGAS 的蛋白序列变化以增强基因组稳定性,为理解衰老的分子演化提供新范式。本研究还通过揭示裸鼹鼠 cGAS–FANCI–RAD50 的分子调控轴,丰富了对 DNA 修复调控网络的认识。此外,本研究成果为开发靶向 cGAS 的 DNA 修复干预疗法奠定基础,提示未来或可通过小分子药物或基因编辑等模拟裸鼹鼠特有变异,为人类对抗衰老及健康长寿开辟新路径。
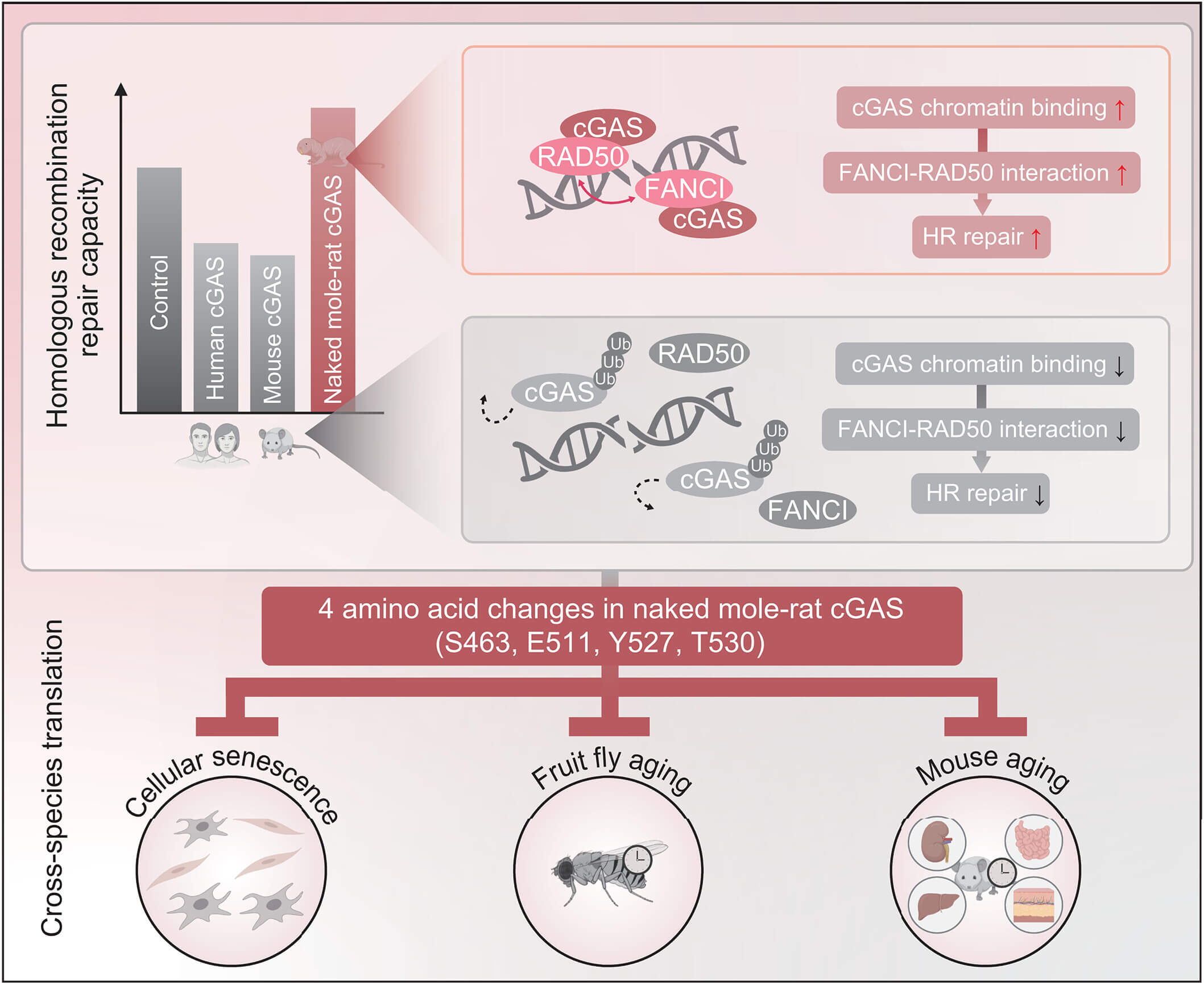
裸鼹鼠 cGAS 促进 HR 修复拮抗衰老
全式金生物产品支撑
优质的试剂是科学研究的利器。全式金生物的 Fast 定点突变试剂盒(FM111)助力本研究。产品自上市以来,凭借优异的性能,深受客户青睐,多次荣登知名期刊,助力科学研究。
Fast Mutagenesis System(FM111)
本产品以甲基化的质粒为模板,采用部分重叠引物 (均含突变点) 设计,使用 2×TransStart® FastPfu Fly PCR SuperMix 扩增,扩增产物用 DMT 限制性内切酶消化甲基化质粒模板后,转化具有降解甲基化质粒模板的感受态细胞。
产品特点
• 由于采用部分重叠引物(均含突变点)设计,使 PCR 呈指数扩增, 扩增产物凝胶电泳可见,扩增产物为环状,易于转化。
• 使用 2×TransStart® FastPfu Fly PCR SuperMix 扩增,缩短了扩增时间,同时提高了扩增的保真性。
• 利用体外限制性内切酶和体内感受态细胞降解甲基化质粒模板,突变效率更高,对照突变效率高达 90%。
使用Fast Mutagenesis System(FM111)产品发表的部分文章:
• Chen C C, Yu Z P, Liu Z W, et al. Chanoclavine synthase operates by an NADPH-independent superoxide mechanism[J]. Nature, 2025.(IF 50.50)
• Zeng Y, Zhang H W, Wu X X, et al. Structural basis of exoribonuclease-mediated mRNA transcription termination[J]. Nature, 2024.(IF 50.50)
• Chen Y, Chen Z X, Wang H, et al. A cGAS-mediated echanism in naked mole-rats potentiates DNA repair and delays aging[J]. Science, 2025.(IF 45.80)
• Liu X, Liu Z, Wu Z, et al. Resurrection of endogenous retroviruses during aging reinforces senescence[J]. Cell, 2023. (IF 45.50)
• Yu Y, Tang W, Lin W, et al. ABLs and TMKs are co-receptors for extracellular auxin[J]. Cell, 2023.(IF 45.50)
• Wang K, Zhang Z, Hang J, et al. Microbial-host-isozyme analyses reveal microbial DPP4 as a potential antidiabetic target[J]. Science, 2023.(IF 44.70)
• Huang K, Wu X X, Fang C L, et al. Pol IV and RDR2: a two-RNA-polymerase machine that produces double-stranded RNA[J]. Science, 2021.(IF 44.70)
• Shi C, Yang X, Hou Y, et al. USP15 promotes cGAS activation through deubiquitylation and liquid condensation[J]. Nucleic Acids Research, 2022.(IF 16.60)
• Wang X, Wang Y, Cao A, et al. Development of cyclopeptide inhibitors of cGAS targeting protein-DNA interaction and phase separation[J]. Nature Communications, 2023.(IF 14.70)
• You L, Shi J, Shen L, et al. Structural basis for transcription antitermination at bacterial intrinsic terminator[J]. Nature communications, 2019.(IF 14.70)
• Tian Y, Chen Z H, Wu P, et al. MIR497HG‐Derived miR‐195 and miR‐497 Mediate Tamoxifen Resistance via PI3K/AKT Signaling in Breast Cancer[J]. Advanced Science, 2023. (IF 14.30)
• Chen K, Sun W, Zhong M, et al. Single-molecule assay guided crRNA optimization enhances specific microRNA detection by CRISPR-Cas12a[J]. Sensors and Actuators B: Chemical, 2024.(IF 8.00)
• Yu C, Xu H, Jiang S, et al. IL-18 signaling is regulated by caspase 6/8 and IL-18BP in turbot (Scophthalmus maximus)[J]. International Journal of Biological Macromolecules, 2024.(IF 7.70)







